Forschungsinteressen
Forschungsprojekte
Ein überspannendes Interesse in unserer Gruppe ist die Evolution von Bakterien und wie sie sich auf wichtige Prozesse, wie die Regulation von Stoffwechselwegen, Genexpression und die Interaktion von Bakterien mit Bakteriophagen auswirkt.
Durch gezielte genetische Veränderungen in den Gram-negativen und Gram-positiven Modelbakterien Escherichia coli und Bacillus subtilis wollen wir herausfinden wie Bakterien sich an Stoffwechsel-Ungleichgewichte anpassen, d.h. wie Selektionsdrücke gezielt genutzt werden können um Bakterien zu isolieren in denen z.B. neue Stoffwechselwege entstanden sind.
So können zum einen völlig neue Stoffwechselwege identifiziert werden und zum anderen bisher unbekannte regulatorische Einflüsse offengelegt werden.
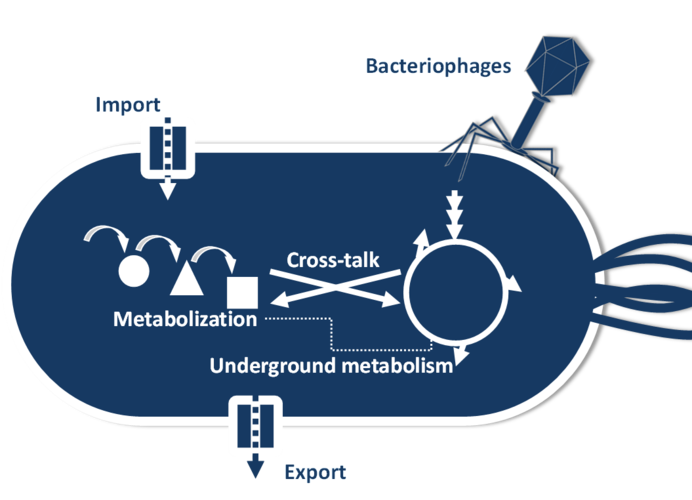
Aktuell interessieren wir uns vor allem für die nähere Charakterisierung eines alternativen Stoffwechselweges, welcher es B. subtilis ermöglicht über einen Fumarat-basierten Stoffwechselweg Ammonium als Stickstoffquelle zu verwerten, um die Aminosäure Glutamat zu synthetisieren (Mardoukhi et al., 2024).
Ein zweiter Themenkomplex ist die Untersuchung der Wechselwirkung zwischen Bakterien und Bakteriophagen, also Viren die spezifisch Bakterien infizieren könnten. Phagen und Bakterien liefern sich seit Millionen von Jahren eine Art „Wettrüsten“ indem Bakterien Verteidigungsmechanismen gegen Phagen-Infektionen entwickeln und die Phagen wiederum Wege finden diese zu umgehen. Die Untersuchung der Koevolution zwischen Bakterien und Phagen erlaubt spannende Einblicke in Prozesse der Evolution und ein besseres Verständnis über die gegenseitige Anpassung von Bakterien und Phagen. Es ist insbesondere essentiell die Resistenzbildungsprozesse zu verstehen um in Zukunft Phagen sicher zur Bekämpfung von pathogenen Bakterien einsetzen zu können.
In diesem Themenkomplex untersuchen wir aktuell ein Restriktions-Modifikationssystem (R-M-System), welches den Bakterien durch kovalente DNA-Modifikationen erlaubt Fremd-DNA von der eigenen DNA zu unterscheiden umso die beispielsweise durch Phagen induzierte Fremd-DNA enzymatisch abbauen zu können. Interessanterweise wird in diesem R-M-System die Genexpression der modifizierenden Methyltransferase (MT) und der DNA-schneidenden Restriktionsendonuklease (RE) durch eine Methylierung des Promoters der beiden für MT und RE kodierenden Gene reguliert. Die Untersuchung dieses R-M-Systems erlaubt es uns daher sowohl ein Abwehrsystem von Bakterien gegenüber Phagen zu charakterisieren, als auch das spannende Thema der Regulation der Genexpression durch DNA-Modifikationen in Bakterien näher zu beleuchten (Beletskaya et al., 2000).
Mardoukhi, M.S.Y., Rapp, J., Irisarri, I., Gunka, K., Link, H., Marienhagen, J., de Vries, J., Stülke, J. & Commichau, F.M. (2024) Metabolic rewiring enables ammonium assimilation via a non-canonical fumarate-based pathway. Microb Biotechnol. 17 (3): e14429.
Beletskaya, I.V., Zakharova, M.V., Shlyapnikov, M.G., Semenova, L.M. & Solonin, A.S. (2000) DNA methylation at the CfrBI site is involved in expression control in the CfrBI restriction-modification system. Nucleic Acids Res. 28 (19): 3817–3822.
