Bakterieller Stoffwechsel & Evolution
Bakterien eignen sich hervorragend zur Untersuchung von Mechanismen und Auswirkungen der Evolution in „Echtzeit“. Durch kurze Generationszeiten und die schiere Anzahl an Zellen kann man eine rasche Divergenz von ursprünglich identischen Zellen beobachten. Wenn Bakterien Selektionsdrücken ausgesetzt sind, d.h. Umwelteinflüssen, die das Überleben der Zellen beeinflussen (Antibiotika, Nährstoffmangel, usw.), ist die erste Anpassungsstrategie i.d.R. eine Änderung der Genexpression. Wenn der Selektionsdruck zu stark ist um das Überleben der Bakterien durch Anpassungen der Genexpression zu erlauben, ist es möglich auf Suppressormutanten zu selektieren, d.h. auf Bakterien, die durch spontane genetische Veränderungen (Genamplifikationen, Punktmutationen, usw.) einen Wachstumsvorteil in Anwesenheit des Selektionsdrucks haben (Sandegren & Andersson, 2009).
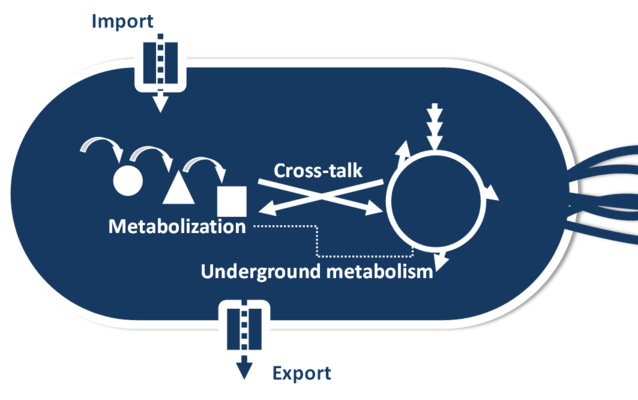
In unseren Forschungen zum bakteriellen Metabolismus machen wir uns eben jene Anpassungsmechanismen der Bakterien zu Nutze, um neue Zusammenhänge in Stoffwechselwegen, deren Regulation und Interkonnektivität zu erleuchten und um gleichzeitig Fragen zur Evolution und Anpassungsfähigkeit von Bakterien zu untersuchen.
Vor kurzem konnte gezeigt werden, dass sowohl Bacillus subtilis, als auch Escherichia coli dazu in der Lage sind unter bestimmten Selektionsdrücken einen alternativen Stoffwechselweg zur Biosynthese der Aminosäure Glutamat durch genetische Veränderungen nutzen können (Mardoukhi et al., 2024; Schulz-Mirbach et al., 2022).
In unserer Forschung setzen wir uns aktuell mit der Frage auseinander wie sich der „klassische“ und der „alternative“ Stoffwechselweg zur Glutamat-Biosynthese unterscheiden, warum die meisten Bakterien den „klassischen“ Stoffwechselweg nutzen und welche Anpassungen der „alternative“ Stoffwechselweg den Bakterien abverlangt.
Mardoukhi, M.S.Y., Rapp, J., Irisarri, I., Gunka, K., Link, H., Marienhagen, J., de Vries, J., Stülke, J. & Commichau, F.M. (2024) Metabolic rewiring enables ammonium assimilation via a non-canonical fumarate-based pathway. Microb Biotechnol. 17 (3): e14429.
Sandegren, L. & Andersson, D.I. (2009) Bacterial gene amplification: implications for the evolution of antibiotic resistance. Nat Rev Microbiol. 7: 578-588.
Schulz-Mirbach, H., Müller, A., Wu, T., Pfister, P., Aslan, S., Schada von Borzyskowski, L., Erb, T.J., Bar-Even, A. & Lindner, S.N. (2022) On the flexibility of the cellular amination network in E coli. eLife. 11: e77492.
